Researchers take a closer look at the genomes of microbial communities in the human mouth
( Harvard University – جامعة هارفارد)
غالبًا ما تُظهر البكتيريا جغرافية حيوية قوية جدًا – بعض البكتيريا موجودة بكثرة في مواقع محددة بينما غائبة عن البعض الآخر – مما يؤدي إلى أسئلة رئيسية عند تطبيق علم الأحياء الدقيقة على العلاجات أو البروبيوتيك: كيف وصلت البكتيريا إلى المكان الخطأ؟ كيف نضيف البكتيريا الصحيحة إلى المكان الصحيح عندما تكون الجغرافيا الحيوية قد خرجت عن السيطرة؟
ومع ذلك، فإن هذه الأسئلة لها عقبة كبيرة واحدة، فالبكتيريا صغيرة جدًا ومتعددة مع مجموعات سكانية متنوعة ومعقدة للغاية مما يخلق تحديات كبيرة لفهم أي مجموعات فرعية من البكتيريا تعيش أين وما هي الجينات أو القدرات الأيضية التي تسمح لها بالنمو في هذه الأماكن “الخطأ”.
وفي دراسة جديدة نُشرت في مجلة أحياء الجينوم (Genome Biology) قام باحثون بقيادة جامعة هارفارد بفحص الميكروبيوم الفموي البشري [١] واكتشفوا تنوعًا مثيرًا للإعجاب في المجموعات السكانية الفرعية البكتيرية التي تعيش في مناطق معينة من الفم.
وقال المؤلف الرئيسي والمرشح لنيل الدكتوراه في قسم البيولوجيا العضوية والتطورية من جامعة هارفارد، دانيال أوتر: “بصفتنا علماء بيئة ميكروبية، نحن مفتونون بكيفية تقسيم البكتيريا على ما يبدو لأي موطن إلى أماكن مختلفة، ولكن كبشر، لدينا أيضًا هذا الفضول الفطري حول كيفية تشكيل الميكروبات داخل أجسامنا”.
وقد قدمت التطورات الأخيرة في التسلسل والنُهُج المعلوماتية الحيوية طرقًا جديدة لفك تعقيد المجتمعات البكتيرية. وتعاون أوتر والبروفيسور كولين كافانو، أستاذ علم الأحياء بقسم الأحياء العضوية والتطورية بجامعة هارفارد، مع باحثين في مختبر الأحياء البحرية في وودز هول، وجامعة شيكاغو، ومعهد فورسيث في كامبريدج بولاية ماساتشوستس، لتطبيق هذه الأساليب الأحدث وأساليب التحليل للحصول على صورة أفضل للميكروبيوم الفموي.
“الفم هو المكان المثالي لدراسة المجتمعات الميكروبية” ، وفقًا للمؤلف المشارك الدكتورة مرات إرين، الأستاذ المساعد في كلية الطب بجامعة شيكاغو. وأضافت الدكتور إرين: “إنها ليست فقط بداية الجهاز الهضمي، ولكنها أيضًا بيئة خاصة جدًا وصغيرة ومتنوعة ميكروبيًا بما يكفي، بحيث يمكننا حقًا البدء في الإجابة عن أسئلة مثيرة للاهتمام حول الميكروبيومات وتطورها”.
ويحتوي الفم على كمية مذهلة من الميكروبات الخاصة بالموقع في مناطق مختلفة. وعلى سبيل المثال، تختلف الميكروبات الموجودة على اللسان اختلافًا كبيرًا عن الميكروبات الموجودة على لوحة الأسنان. وقالت الدكتورة إرين: “ميكروبات لسانك تشبه تلك التي تعيش على لسان شخص آخر أكثر من تلك التي تعيش في حلقك أو على لثتك!”.
وقد قام الفريق بتفتيش قواعد البيانات العامة وتنزيل ١٠٠ جينوم تمثل أربعة أنواع من البكتيريا الموجودة عادة في الفم، هيموفيلوس باراينفلونزاي (Haemophilus parainfluenzae) والأنواع الثلاثة الفموية من جنس روثيا (Rothia) ، واستخدموها كمراجع للتحقيق في عينات مشابهة في مئات أفواه المتطوعين من مشروع الميكروبيوم البشري. وقال أوتر: “لقد استخدمنا هذه الجينومات كنقطة انطلاق، لكن سرعان ما تجاوزناها لبحث التباين الجيني الكلي بين تريليونات الخلايا البكتيرية التي تعيش في أفواهنا” ، وتابع: “لأنه في نهاية المطاف، هذا ما يثير فضولنا، وليس القلة الاعتباطية التي تم ترتيبها بشكل متسلسل”.
وباستخدام هذا النهج الذي تم تطويره مؤخرًا والمسمى ميتابانجينومكس (metapangenomics) ، والذي يجمع بانجينوم (pangenomes) (مجموع كل الجينات الموجودة في مجموعة من البكتيريا ذات الصلة) مع ميتاجينومكس (metagenomics) (دراسة الحمض النووي الكلي القادم من جميع البكتيريا في المجتمع) ، سمح للباحثين بإجراء الفحص المتعمق لجينوم الميكروبات مما أدى إلى اكتشاف صادم. وقال أوتر: “لقد وجدنا قدرًا هائلاً من التباين، لكننا شعرنا بالصدمة من نمط هذا التباين عبر أجزاء مختلفة من الفم؛ على وجه التحديد، بين اللسان والخد وأسطح الأسنان”.
وعلى سبيل المثال، وجد الباحثون، داخل نوع واحد من الميكروبات، أشكالًا جينية مميزة مرتبطة بقوة بموقع واحد مختلف داخل الفم. وفي كثير من الحالات، تمكن الفريق من تحديد عدد قليل من الجينات التي قد تفسر موطن مجموعة بكتيرية معينة. وبتطبيق علم الميتابانجينومكس، تمكن الباحثون أيضًا من تحديد طرق معينة تختلف فيها البكتيريا الحية الحرة في أفواه الناس عن المماثلين لها الذين نمو في المختبر.
ويشير الدكتور كافانو إلى أن “الدقة التي توفرها هذه التقنيات – من خلال المقارنة المباشرة لجينومات البكتيريا “المدجنة” و”الجامحة”- تسمح لنا بتشريح هذه الاختلافات الجينية حسب الجين”. وقال: “تمكنا أيضًا من تحديد سلالات بكتيرية جديدة مرتبطة بالسلالات الموجودة في الاستنبات، ولكنها تختلف عنها”.
وأضاف الدكتور كافانو: “بعد تحديد بعض البكتيريا المرشحة القوية حقًا التي يمكنها تحديد التكيف مع موطن معين، نود اختبار هذه الفرضيات بشكل تجريبي”. ومن المحتمل أن تكون هذه النتائج هي المفتاح لفتح لاطلاق البروبيوتيك المستهدف، حيث يمكن للعلماء استخدام ما تم تعلمه حول متطلبات موطن كل ميكروب لهندسة الميكروبات المفيدة للهبوط في موطن معين.
وقالت المؤلفة المشاركة جيسيكا مارك ويلش، العالمة المشاركة في المختبر البيولوجي البحري: “يسهل الوصول إلى الفم لدرجة أن الناس كانوا يعملون على البكتيريا من الفم لفترة طويلة”. وأضافت: “كل بيئة ننظر إليها تحتوي على هذه المجتمعات المعقدة من البكتيريا، ولكن لماذا هذا؟”. وتابعت: “إن فهم سبب تعقيد هذه المجتمعات وكيفية تفاعل البكتيريا المختلفة سيساعدنا على فهم أفضل لكيفية إصلاح مجتمع بكتيري يضر بصحتنا، ويخبرنا بالميكروبات التي يجب إزالتها أو إضافتها مرة أخرى”.
ويمكن أن توفر هذه الدراسة وغيرها من الدراسات المشابهة رؤى جديدة حول دور ميكروبات الفم في صحة الإنسان. وقال أوتر: “إن القدرة على تحديد جينات معينة وراء تكيف السكان كانت إلى حد ما “الكأس المقدسة” في البيئة الميكروبية، ونحن متحمسون للغاية لمساهماتنا في هذا المجال!”
*تمت الترجمة بتصرف
لمزيد من المعلومات: دانيال اوتر وآخرين ؛ Metapangenomics of the oral microbiome provides insights into habitat adaptation and cultivar diversity, Genome Biology (2020). DOI: 10.1186/s13059-020-02200-2
المصدر:
https://phys.org/news/2020-12-closer-genomes-microbial-human-mouth.html
الهوامش:
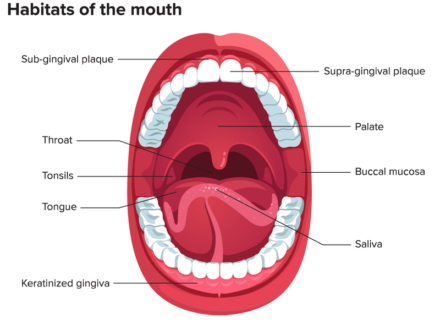
[١] يحتوي تجويف الفم البشري على عدد من السكان المختلفين، بما في ذلك الأسنان، والتلم اللثوي، واللسان، والخدين، والحنك الصلب واللين، واللوزتين، التي تستعمرها البكتيريا. يتكون الميكروبيوم الفموي من أكثر من 600 نوع سائد على مستوى الأصناف، مع مجموعات فرعية مميزة تسود في السكان المختلفة. تم تمييز الميكروبيوم الفموي على نطاق واسع من خلال الرعاية وطرق الاستنبات المستقلة الجزيئية مثل استنساخ الرنا الريباسي١٦س (16S nRNA).
 علوم القطيف مقالات علمية في شتى المجالات العلمية
علوم القطيف مقالات علمية في شتى المجالات العلمية